对比学习和专家混合可在生物数据库中实现精确的向量嵌入
快速阅读: 据《Nature.com》称,研究通过引入专家混合(MoE)框架改进生物医学领域文本向量嵌入,利用共引作为相似性度量优化预训练BERT模型,实现高效编码异构输入,提升向量数据库检索与整合效率,且保持与常规Transformer相同吞吐量。
Transformer神经网络的发展显著提升了句子相似性模型的性能,然而,这些模型在高度区分性的任务上往往表现不佳,并且难以生成复杂文档(如同行评审的科学文献)的最佳表示。随着对检索增强和搜索的依赖不断增加,将结构和主题各异的研究文档表示为简洁而具有描述性的向量显得尤为重要。
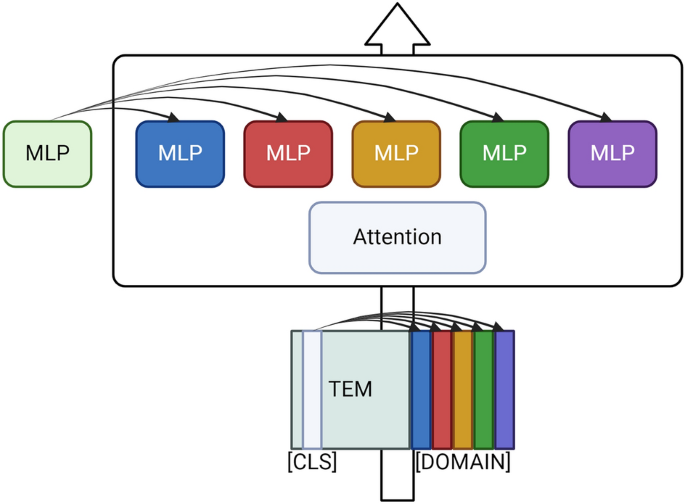
本研究通过利用共引作为相似性度量,构建了面向生物医学领域的特定数据集,改进了科学文本的向量嵌入方式。我们提出了一种新颖的专家混合(MoE)扩展框架,应用于预训练的BERT模型。其中,每个多层感知器模块被复制给不同的专家。我们的MoE变体经过训练,能够根据多个生物学领域的科学摘要判断两个出版物是否在另一篇论文中共同被引用(共引)。值得一提的是,由于我们采用了基于特殊标记的独特路由机制,扩展后的MoE系统吞吐量与常规Transformer保持一致。这为构建用于编码异构生物医学输入的一体化Transformer网络奠定了基础。
我们的方法推动了表征学习的发展,并有望提升向量数据库的检索和整合效率。
(以上内容均由Ai生成)